2016年10月27日,国际著名学术期刊Molecular Cell在线发表了中国科学院上海生命科学研究院生物化学与细胞生物学研究所陈玲玲研究组关于长非编码RNA的最新进展“Unusual Processing Generates SPA LncRNAs that Sequester Multiple RNA Binding Proteins”。该研究发现一类新型长非编码RNA,揭示其加工产生机制及参与剪接调控的重要功能机制。
众所周知,人类基因组中存在大量被称为基因组“暗物质(dark matter)”的非编码序列,包括基因间非编码序列、内含子非编码序列等。基因间非编码序列转录产生的线形长非编码RNA(lncRNA)在分子形式上与信使RNA (mRNA)类似,具有5'端m7G帽子和3'poly(A)尾巴的结构。m7G帽子结构对RNA稳定性、成熟以及出核等至关重要。
陈玲玲实验室前期发现了内含子来源的两端以小核仁RNA(snoRNA)结尾的lncRNA,命名为sno-lncRNAs(Yin et al., Mol Cell, 2012, Cover Story, Best of Mol Cell 2012)。为寻找与snoRNA相关的新型lncRNA分子家族,该研究通过RNA免疫共沉淀Box C/D类型的snoRNP复合物核心蛋白Fibrilarin(Fib-RIP)并结合高通量测序发现了一类新类RNA分子。这类长非编码RNA的5'端是snoRNA帽子而非经典的m7G帽子,3'端是poly(A)尾巴,并经过多个外显子剪接加工,我们命名这类新RNA分子为SPA(5'snoRNA capped and 3'polyadenylatedlncRNAs)。研究揭示SPA的加工产生依赖上游较弱的转录终止信号、5'端snoRNA帽子、3' processing 因子CPSFs、XRN2降解pre-RNA以及较快的Pol II延伸速度(如图)。
其中两条SPAs来源于15号染色体基因印记q11-q13区域编码基因SNRPN的3' UTR区域。而该区域仅在父系来源的染色体表达,并在几乎所有的小胖威利综合症(Prader-Willi Syndrome,PWS)的患者中完全缺失或不被转录。值得一提的是,之前发现的两端以小核仁RNA结尾的五条sno-lncRNAs也是位于该PWS关键缺失区域(Yin et al., Mol Cell, 2012)。功能研究表明,SPAs与sno-lncRNAs在每一个正常的人源胚胎干细胞(hESC)和正常人来源的诱导多功能干细胞(iPSC)内均形成一个1 ~2 μm3的细胞核内积聚小体(如图)。在此核积聚小体内,SPAs与sno-lncRNAs结合了大量的可变剪接调控蛋白TDP43、RBFOX2、hnRNP M。利用CRISPR/CAS9技术在hESCs中将父系来源的染色体包含SPAs与sno-lncRNAs的区域敲除,制备PWS疾病的人源胚胎干细胞模型。通过全转录组RNA测序和单核苷酸分辨率交联和免疫共沉淀法(individual-nucleotide resolution UV cross-linking and immunoprecipitation,iCLIP)分析,进一步发现这些PWS关键区域lncRNAs的缺失改变了TDP43、RBFOX2、hnRNP M等与一些pre-mRNA的结合,进而影响了可变剪接。有意思的是,SPAs与sno-lncRNAs对TDP43和RBFOX2的并结合具有偏好性,并进而可以调控相应的pre-mRNA可变剪接。
值得注意的是,PWS是一类基因疾病,该疾病病理机制至今不详,表型主要为神经系统发育异常、激素分泌失衡、肥胖、智力低下等。PWS病人中15号染色体q11-q13区域基因表达缺失,最小核心缺失区域为108kb。正常情况下,SPAs、sno-lncRNAs等长非编码RNA均产生自该PWS核心缺失区域,从而提示这些RNA分子的表达缺失可能与PWS综合症的发生发展密切相关。该工作为进一步了解PWS综合症的病理机制提供了新的研究思路。
该项研究在陈玲玲研究员的指导下,主要由生化细胞所博士研究生吴煌,博士后殷庆飞,计算生物学研究所博士研究生罗政共同完成,同时该研究还得到了计算生物学研究所杨力研究员的大力支持。该课题得到了国家科技部和基金委的经费支持。
文章链接:http://www.cell.com/molecular-cell/fulltext/S1097-2765(16)30630-X
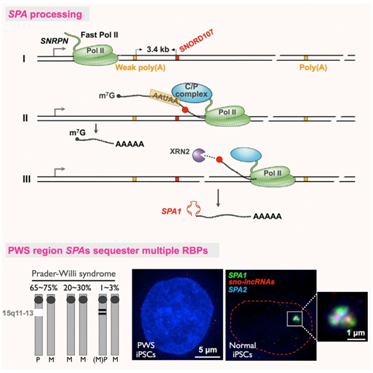
SPA加工产生机制模型(上图)及PWS区域lncRNAs在iPS细胞(PWS病人来源、正常人来源)中定位(下图)。